Realtime PCR
앞서 realtime PCR에 대해 여러 포스팅으로 알아봤는데요! 오늘은 원하는 샘플의 유전자 발현이 어느 정도로 나타났는지 확인하는 방법에 알아보겠습니다. 우선 종류들이 어떤 것인지 말씀드리고 예시를 통해 이해하기 쉽게 설명드리도록 할게요!
종류
실시간 PCR의 중요한 측면 중 하나는 대상 유전자 발현에서 관찰된 변화가 시작 물질의 양이나 품질의 차이로 인한 것이 아님을 보장하기 위해 데이터를 정규화하는 것이에요. 정규화는 서로 다른 샘플에서 안정적으로 발현되는 하나 이상의 참조 유전자(하우스키핑 유전자라고도 함)의 발현을 측정하여 확인하죠.
1. 상대정량화: 표적 유전자를 참조 유전자와 비교하는 방법이에요. 참조유전자를 놓고 상대적으로 얼마나 유전자가 발현이 되었는지를 확인할 수 있죠. 위의 House keeping gene 도 이에 해당하는 방법이에요.
2. 절대정량화: 표적 유전자의 표준곡선을 통해서 정량화를 진행하는 방법이에요. 각 샘플에서 표적 유전자의 절대량을 결정하게 되죠. 표준곡선을 이용해 정량화하는 만큼 샘플들의 PCR 효율이 일정하게 같은 효율이 나온다고 생각하는 경우 시행하게 됩니다.
3. ∆Ct법: 이 방법은 대상유전자와 참조 유전자 사이의 Ct값의 차이를 계산하고 ∆Ct 값을 이용해서 표적유전자의 상대적 발현을 계산하면 됩니다. 단순히 대상 유전자와 참조 유전자사이의 발현량 차이를 확인하는 방법으로 실제 샘플의 PCR 효율은 고려되지 않은 단순한 ∆Ct 값만 비교하는 방법이에요.
4. ∆∆Ct법: 테스트샘플과 대조 샘플의 ∆Ct값 차이를 계산하고 ∆∆Ct값을 이용해서 표적유전자 발현의 배수 변화를 계산하는 방법이에요. 모든 유전자 내에 존재하는 housekeeping gene을 이용하여 발현을 확인하고 이 값을 이용하여 테스트 샘플의 발현량을 보정하여 각 샘플마다 나타나는 발현량의 차이를 없애 보다 정확한 결괏값을 낼 수 있어요.
계산 방법
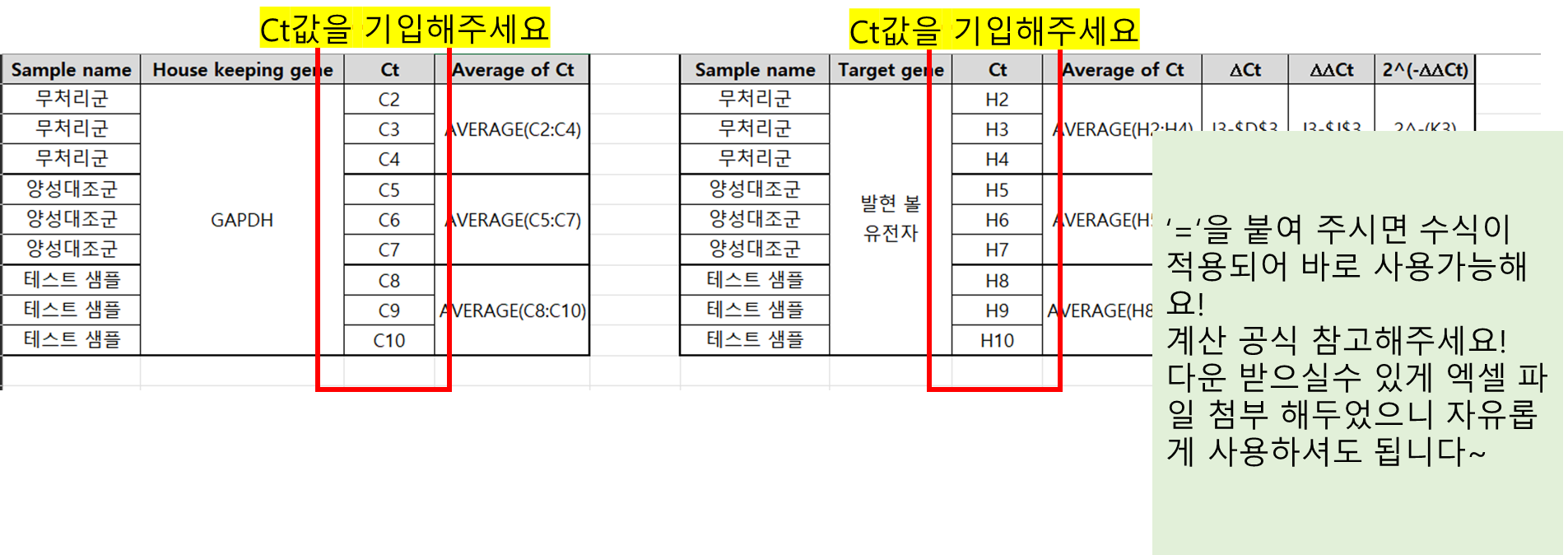
1. realtime PCR후 나타나는 Ct값의 평균을 구해줍니다.
2. 평균을 구한 후 ∆Ct는 발현을 보고자 하는 유전자의 Ct값 평균 - housekeeping gene 유전자(GAPDH등)의 Ct 평균을 하면 값이 나오게 됩니다.(발현유전자 Ct 평균 - GAPDH Ct 평균)
3. 무처리군 대비 양성 샘플의 ∆Ct값을 확인하면 ∆∆Ct값이 구해지게 됩니다.(양성 샘플 ∆Ct - 무처리군 샘플 ∆Ct)
4. 3번에서 구해진 결과(∆∆Ct)를 2^(-∆∆Ct)에 대입한 후 결과를 구해주면 정확한 발현량의 차이를 log2-배수차이로 확인할 수 있습니다.
조금 더 이해 하기 쉬우시도록 엑셀로 정리해 봤어요 필요하신 분들 참고해 주세요~
엑셀 안에 = 기호 넣으면 되시고, Ct값은 기입해 주시면 됩니다.
'실험 정보' 카테고리의 다른 글
| [실험 정보] 실험 기초 정복 몰 농도 계산(개념, 공식, 예시) (0) | 2023.03.27 |
|---|---|
| [실험 정보] 실험 기초 정복 Serial dilution (0) | 2023.03.26 |
| [실험 정보] Real-time PCR 용어 정리(feat. 실험 안될 때 해결 방법) (0) | 2023.03.16 |
| [실험 정보] 세포 동결보존(Cell Freezing)준비물, 실험방법, 주의사항 (1) | 2022.06.26 |
| [실험 정보] API Test(20E, 20NE), 그람음성균에 대한 생화학적 시험 (1) | 2022.06.24 |